Тетратрикопептидный повтор - Tetratricopeptide repeat
| Тетратрикопептидный повтор | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| Идентификаторы | |||||||||||
| Символ | TPR_1 | ||||||||||
| Pfam | PF00515 | ||||||||||
| Pfam клан | CL0020 | ||||||||||
| ИнтерПро | IPR001440 | ||||||||||
| SCOP2 | 1a17 / Объем / СУПФАМ | ||||||||||
| CDD | cd00189 | ||||||||||
| |||||||||||
В тетратрикопептидный повтор (TPR) это структурный мотив. Он состоит из выродиться 34 аминокислоты тандемный повтор выявлен в большом количестве белки. Встречается в тандемных массивах из 3–16 мотивов,[1] которые формируют каркасы для обеспечения белок-белковых взаимодействий и часто сборки мультибелковых комплексов. Эти альфа-спираль пара повторений обычно складывать вместе для создания единого линейного соленоидный домен называется TPR домен. Белки с такими доменами включают комплекс, способствующий анафазе (APC) субъединицы cdc16, cdc23 и cdc27, то НАДФН оксидаза подразделение p67-phox, связывание hsp90 иммунофилины, факторы транскрипции, то протеинкиназа R (PKR), главный рецептор импорта белков пероксисомального матрикса PEX5, протеин-аргининметилтрансфераза 9 (PRMT9) и митохондриальные импортные белки.

Структура
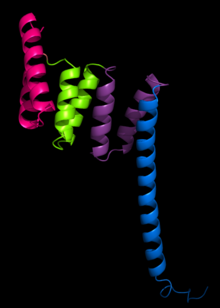
Структура Белок PP5 была первой структурой, которую нужно было определить. Структура решена Рентгеновская кристаллография Das и его коллеги показали, что мотив последовательности TPR состоит из пары антипараллельных альфа-спиралей.[2] Структура PP5 содержала 3 тандемных повтора TPR, которые показали, что последовательные повторы TPR образуют альфа-спираль. соленоид структура.
Типичная структура TPR характеризуется взаимодействиями между спиралями A и B первого мотива и спиралью A ’следующего TPR. Хотя природа таких взаимодействий может варьироваться, первые две спирали TPR-мотива обычно имеют угол упаковки ~ 24 градусов в пределах одного мотива. Повторы более чем трех мотивов TPR создают правую суперспираль, характеризующуюся как вогнутой, так и выпуклой гранью, из которых вогнутая сторона обычно участвует в связывании лиганда.[1] [3]

Что касается последовательности, TPR содержит смесь малых и больших гидрофобных остатков, тем не менее, никакие положения не являются полностью инвариантными. Однако есть некоторые остатки, которые обычно консервативны, включая триптофан 4, лейцин 7, глицин 8, тирозин 11, аланин 20, фенилаланин 24, аланин 27 и пролин 32. Среди этих 8 аланина в положениях 8, 20 и 27 обычно больше. сохранено. В других позициях более предпочтительны небольшие, большие или ароматические аминокислоты, чем конкретный остаток. Между спиралями сохранение остатков играет больше структурную роль, поскольку присутствуют остатки, разрушающие спираль. Между соседними TPR остатки играют как структурные, так и функциональные роли.[1]
TPR содержащие пептиды
Прыгать
В Прыгать адаптерный белок опосредует ассоциацию молекулярных шаперонов Hsp70 и Hsp90. Он содержит три 3-TPR-повтора, каждый со своей специфичностью связывания пептидов. Известно, что его домен TPR1 распознает C-конец Hsp70, в то время как TPR2 связывается с C-концом Hsp90. Обе С-концевые последовательности заканчиваются мотивом EEVD, и природа взаимодействия является как электростатической, так и гидрофобной.[1][4]
PEX5
В PEX5 белок является рецептором для PTS1 (трипептид пероксисомального нацеленного сигнала, который направляет белки в пероксисомы). Он взаимодействует с сигналом через мотивы TPR. Большинство его контактов с C-концевым трипептидом PTS1 находится на вогнутой поверхности TPRs 1, 2 и 3.[5]
Цитозольный фактор нейтрофилов 2
Цитозольный фактор нейтрофилов 2 является важным элементом комплекса НАДФН-оксидазы, который, в свою очередь, производит супероксиды в ответ на микробную инфекцию. Связывание Rac GTPase является ключевым шагом в сборке комплекса, и TPR в модуле phox опосредуют сборку мультибелкового комплекса, действуя как связывающий каркас.[6]
Примеры
Человеческие гены, кодирующие белки, содержащие этот мотив, включают:
- AAG2, ANAPC7
- BBS4
- КАБИНА1, CDC16, CDC23, CDC27, CNOT10, CTR9
- DNAJC3, DNAJC7, DYX1C1
- FAM10A4, FAM10A5, FKBP4, FKBP5, FKBP8, FKBPL
- GPSM1, GPSM2, GTF3C3
- IFIT1, IFIT1L, IFIT2, IFIT3, IFIT5, IFT140, IFT88
- KLC1, KLC2, KLC3, KLC4, KNS2
- LONRF2
- NARG1, NARG1L, НАСП, NCF2, NFKBIL2, NOXA1, НПХП3
- OGT
- PEX5, PEX5L, PPID, PPP5C, PRPF6
- РАНБП2, RANBP2L2, RANBP2L6, РАПСН, RGPD5, RGPD7, RPAP3
- SGTA, SGTB, SH3TC1, SH3TC2, SPAG1, SRP72, ST13, STIP1, STUB1, SUGT1
- TMTC1, TMTC2, TMTC3, TMTC4, TOMM34, TOMM70A
- TTC1, TTC3, TTC4, TTC5, TTC6, TTC7A, TTC7B, TTC8, TTC9C, TTC12, TTC13, TTC14, TTC15, TTC16, TTC17, TTC18, TTC21A, TTC21B, TTC22, TTC24, TTC25, TTC27, TTC28, TTC29, TTC30A, TTC30B, TTC31, TTC33, TTC37
- UNC45A, UNC45B, UTX, UTY
- WDTC1
- ZC3H7B
Рекомендации
- ^ а б c d Blatch GL, Lässle M (ноябрь 1999 г.). «Тетратрикопептидный повтор: структурный мотив, опосредующий белок-белковые взаимодействия». BioEssays. 21 (11): 932–9. Дои:10.1002 / (SICI) 1521-1878 (199911) 21:11 <932 :: AID-BIES5> 3.0.CO; 2-N. PMID 10517866.
- ^ Дас А.К., Коэн П.В., Барфорд Д. (март 1998 г.). «Структура тетратрикопептидных повторов протеинфосфатазы 5: значение для межбелковых взаимодействий, опосредованных TPR». Журнал EMBO. 17 (5): 1192–9. Дои:10.1093 / emboj / 17.5.1192. ЧВК 1170467. PMID 9482716.
- ^ Уилсон CG, Kajander T, Regan L (январь 2005 г.). «Кристаллическая структура NlpI. Прокариотический тетратрикопептидный повторяющийся белок с глобулярной складкой». Журнал FEBS. 272 (1): 166–79. Дои:10.1111 / j.1432-1033.2004.04397.x. PMID 15634341.
- ^ Шойфлер С., Бринкер А., Буренков Г., Пегораро С., Мородер Л., Бартуник Х., Хартл Ф.Ю., Моарефи I (апрель 2000 г.). «Структура комплексов TPR домен-пептид: критические элементы в сборке многочапероновой машины Hsp70-Hsp90». Клетка. 101 (2): 199–210. Дои:10.1016 / S0092-8674 (00) 80830-2. PMID 10786835. S2CID 18200460.
- ^ Gatto GJ, Geisbrecht BV, Gould SJ, Berg JM (декабрь 2000 г.). «Распознавание пероксисомального нацеленного сигнала-1 TPR-доменами человеческого PEX5». Структурная биология природы. 7 (12): 1091–5. Дои:10.1038/81930. PMID 11101887. S2CID 35168630.
- ^ Lapouge K, Smith SJ, Walker PA, Gamblin SJ, Smerdon SJ, Rittinger K (октябрь 2000 г.). «Структура TPR домена p67phox в комплексе с Rac.GTP». Молекулярная клетка. 6 (4): 899–907. Дои:10.1016 / S1097-2765 (05) 00091-2. PMID 11090627.
дальнейшее чтение
- Лима Мде Ф., Элой Н.Б., Пегораро С., Сагит Р., Рохас С., Бретц Т., Варгас Л., Элофссон А., де Оливейра А.С., Хемерли А.С., Феррейра П.С. (18 ноября 2010 г.). «Геномная эволюция и сложность комплекса, стимулирующего анафазу (APC) наземных растений». BMC Биология растений. 10: 254. Дои:10.1186/1471-2229-10-254. ЧВК 3095333. PMID 21087491.
- Дас А.К., Коэн П.В., Барфорд Д. (март 1998 г.). «Структура тетратрикопептидных повторов протеинфосфатазы 5: значение для межбелковых взаимодействий, опосредованных TPR». Журнал EMBO. 17 (5): 1192–9. Дои:10.1093 / emboj / 17.5.1192. ЧВК 1170467. PMID 9482716.
- Whitfield C, Mainprize IL (февраль 2010 г.). «Мотивы TPR: отличительные черты нового экспортного каркаса полисахаридов». Структура. 18 (2): 151–3. Дои:10.1016 / j.str.2010.01.006. PMID 20159460.
- Крахлер AM, Шарма A, Kleanthous C (июль 2010 г.). «Самоассоциация доменов TPR: уроки, извлеченные из разработанного, основанного на консенсусе олигомера TPR». Белки. 78 (9): 2131–43. Дои:10.1002 / prot.22726. PMID 20455268.
- Schapire AL, Valpuesta V, Botella MA (сентябрь 2006 г.). «Белки TPR в передаче сигналов гормонов растений». Сигнализация и поведение растений. 1 (5): 229–30. Дои:10.4161 / psb.1.5.3491. ЧВК 2634123. PMID 19704665.
- Cortajarena AL, Regan L (май 2006 г.). «Связывание лиганда TPR доменами». Белковая наука. 15 (5): 1193–8. Дои:10.1110 / пс. 062092506. ЧВК 2242500. PMID 16641492.
- Д'Андреа Л.Д., Риган Л. (декабрь 2003 г.). «Белки TPR: универсальная спираль». Тенденции в биохимических науках. 28 (12): 655–62. CiteSeerX 10.1.1.313.8712. Дои:10.1016 / j.tibs.2003.10.007. PMID 14659697.
- Гёбл М., Янагида М. (май 1991 г.). «Snap Helix TPR: новый мотив повтора белка от митоза до транскрипции». Тенденции в биохимических науках. 16 (5): 173–7. Дои:10.1016 / 0968-0004 (91) 90070-C. PMID 1882418.
внешняя ссылка
- Ресурс Eukaryotic Linear Motif класс мотива LIG_TPR
- Ресурс Eukaryotic Linear Motif класс мотива TRG_PTS1
