Комбинированный бисульфитный рестрикционный анализ - Combined bisulfite restriction analysis
Комбинированный анализ ограничения бисульфита (или же КОБРА) - это метод молекулярной биологии, который позволяет проводить точную количественную оценку Метилирование ДНК уровни в конкретном геномном локусе на Последовательность ДНК в небольшом образце геномной ДНК.[1] Техника представляет собой разновидность бисульфитное секвенирование, и сочетает преобразование бисульфита основан полимеразной цепной реакции с ограничение пищеварения. Первоначально разработан для надежной обработки небольших количеств геномной ДНК из микродиссекций. парафин -внедренные образцы тканей, метод с тех пор широко используется в исследования рака и эпигенетика исследования.[2]
Процедура
Бисульфитная обработка
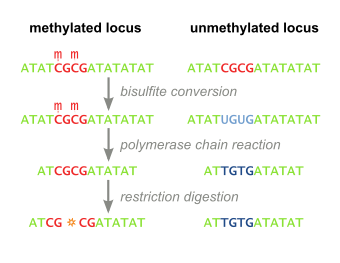
Интересующая геномная ДНК обрабатывается бисульфит натрия, который вводит различия в последовательности, зависящие от метилирования. Во время лечения бисульфитом натрия неметилированные цитозин остатки превращаются в урацил, пока метилированный цитозин остатки не затронуты.

ПЦР-амплификация

Обработанная бисульфитом ДНК затем амплифицируется с помощью ПЦР, что приводит к остаткам цитозина в исходно метилированных положениях и остаткам тимина в исходно неметилированном положении (которые были преобразованы в урацил). Праймеры, используемые на этом этапе, не содержат CpG сайты (общая мишень метилирования цитозина), поэтому процесс амплификации не делает различий между матрицами на основе статуса метилирования. Продукты ПЦР очищенный чтобы обеспечить полное пищеварение на следующем этапе.
Ограничительный дайджест
Вышеуказанные шаги приводят к зависимому от метилирования удержанию или потере CpG-содержащих сайты рестрикционных ферментов, например, для TaqI (TCGA) и BstUI (CGCG), в зависимости от того, был ли остаток цитозина изначально метилирован или нет, соответственно. Из-за независимой от метилирования амплификации на вышеуказанном этапе полученные продукты ПЦР будут представлять собой смешанную популяцию фрагментов, которые потеряли или сохранили сайты рестрикционных ферментов, содержащих CpG, соответствующие процентные доли которых будут напрямую коррелировать с исходным уровнем метилирования ДНК в образец ДНК.
Затем продукты ПЦР обрабатывают рестрикционным ферментом (например BstUI), который будет расщеплять только сайты, которые изначально были метилированы (CGCG), оставляя сайты, которые изначально были неметилированы (TGTG). Чтобы гарантировать, что все сайты CpG сохраняются из-за исходного метилирования, а не остатки неполного превращения бисульфита, проводится контрольное расщепление с помощью таких ферментов, как Hsp92II который распознает последовательность CATG, ни одна из которых не должна оставаться после превращения бисульфита (за редким исключением метилирования не-CpG), и, таким образом, расщепление не должно происходить, если превращение бисульфита было полным.
Количественная оценка
Затем переваренные фрагменты разделяются электрофорез в полиакриламидном геле с ожидаемым появлением полос, соответствующих одному большому непереваренному фрагменту, и множества более мелких полос, соответствующих расщепленным фрагментам. Количественное количество ДНК в этих полосах можно определить с помощью такого устройства, как фосфоимиджер, после чего процент метилирования исходного образца можно рассчитать следующим образом:
Использование и приложения
COBRA широко использовалась во многих исследовательских приложениях, таких как скрининг изменений метилирования ДНК в промоторы генов в исследованиях рака,[3][4] обнаружение измененных паттернов метилирования в импринтированные гены,[5] и характеристика паттернов метилирования в геноме во время развития у млекопитающих.[6][7]
В медицине КОБРА использовалась как инструмент для диагностики заболеваний человека, связанных с аберрантным метилированием ДНК. Исследователи использовали COBRA в сочетании с денатурирующая высокоэффективная жидкостная хроматография в диагностике генетический импринтинг беспорядок Синдром Рассела-Сильвера куда гипометилирование импринтированного гена H19 вызывает нарушение у 50% пациентов.[8]
Сильные стороны
- Просто, быстро и недорого: В COBRA уровни метилирования ДНК легко и быстро измеряются без необходимости трудоемкого субклонирования и последовательность действий, как и в случае бисульфитного секвенирования. Анализ прост и может проводиться с использованием стандартных недорогих реагентов молекулярной биологии.
- Высокая совместимость: Благодаря стадиям ПЦР и очистки, метод работает не только с очень небольшими количествами геномной ДНК, но и с образцами, обработанными парафином, что может быть проблемой в других протоколах количественного определения метилирования ДНК, таких как Саузерн-блоттинг и расщепление чувствительным к метилированию рестрикционным ферментом с последующей ПЦР.
- Количественный: Это в отличие от ПЦР, специфичная для метилирования, что качественно. С помощью COBRA уровни метилирования ДНК могут быть непосредственно определены количественно в данном локусе, что дает больше информации для каждого анализа.
- Масштабируемость для высокопроизводительной обработки проб: С COBRA многие интересующие области могут обрабатываться параллельно в отдельных образцах, обработанных одним и тем же рестрикционным ферментом. Это контрастирует с анализом бисульфитного секвенирования, где необходимо тщательно исследовать каждую область путем секвенирования множества клонов на локус, что требует больше времени.
- Несколько запросов на анализ: Статус метилирования может быть исследован на нескольких сайтах рестрикции, содержащих CpG, в одном анализе переваривания.
Недостатки
- Анализ ограничено использованием существующих ограничительных сайтов в интересующей области, и метилирование, которое не происходит в контексте конкретного сайта рестрикции, не будет анализироваться.
- Неполное пищеварение рестрикционными ферментами после ПЦР может затруднить анализ: неполное переваривание может свидетельствовать об отсутствии метилирования ДНК (при разрезании чувствительным к метилированию ферментом, таким как HpaII). Также известно, что BstUI может разрезать неконвертированные сайты, что приводит к завышенной оценке уровней метилирования, поэтому часто требуется использование HpaII.[9]
- В сложных образцах неоднородность клеточного типа может затруднить анализ поскольку ДНК не секвенируется, неоднородность последовательностей из разных клеток в образце (т.е. различные популяции клеток в опухоли), которые приобрели мутации в исследуемой области, такие как изменение динуклеотида CG на CA или CT, может привести к потере сайта рестрикции, что приведет к появлению явно метилированной области из-за недостаточного переваривания. Это исказило бы количественную оценку уровней метилирования ДНК в данном образце.
Альтернативы
Как правило, COBRA часто сочетается с другими анализами метилирования ДНК и часто используется при первоначальном скрининге интересующих локусов. Если COBRA предполагает изменение паттернов метилирования, тогда могут быть применены более строгие и трудоемкие методы, такие как бисульфитное секвенирование или MeDIP. Также для обнаружения метилирования ДНК можно использовать секвенирование PacBio.
Рекомендации
- ^ Сюн, Чжэнган; Лэрд, Питер В. (1997). «КОБРА: чувствительный и количественный анализ метилирования ДНК». Исследования нуклеиновых кислот. 25 (12): 2532–2534. Дои:10.1093 / nar / 25.12.2532. ЧВК 146738. PMID 9171110.
- ^ Лэрд, Питер У. (2003). «Сила и перспективы маркеров метилирования ДНК». Обзоры природы Рак. 3 (Апрель): 253–266. Дои:10.1038 / nrc1045. PMID 12671664. По данным на 2–11 февраля, на эту статью было обращено 576 ссылок. Scopus
- ^ Шен, Ланлань; и другие. (2005). «Метилирование промотора MGMT и дефект поля при спорадическом колоректальном раке». Журнал Национального института рака. 97 (18): 1330–1338. CiteSeerX 10.1.1.536.6096. Дои:10.1093 / jnci / dji275. PMID 16174854.
- ^ Сутер, Кэтрин, М .; и другие. (2004). «Эпимутация зародышевой линии в MLH1 у людей с множественными раками». Природа Генетика. 36 (5): 497–501. Дои:10,1038 / ng1342. PMID 15064764.
- ^ Блик, Джет; и другие. (2009). «Гипометилирование множественных материнских метилированных импринтированных областей, включая локусы PLAGL1 и GNAS в синдроме Беквита – Видеманна, и характеризация паттернов метилирования ДНК в геноме во время развития у млекопитающих» (PDF). Европейский журнал генетики человека. 17 (5): 611–618. Дои:10.1038 / ejhg.2008.233. ЧВК 2986258. PMID 19092779.
- ^ Верниг, Мариус; и другие. (2007). «Перепрограммирование фибробластов in vitro в плюрипотентное состояние, подобное ES-клеткам». Природа. 448 (7151): 318–324. Bibcode:2007 Натур.448..318Вт. Дои:10.1038 / природа05944. PMID 17554336.
- ^ Mikkelsen, Tarjei S .; и другие. (2008). «Рассечение прямого репрограммирования с помощью интегративного геномного анализа». Природа. 454 (Июль): 49–56. Bibcode:2008Натура.454 ... 49М. Дои:10.1038 / природа07056. ЧВК 2754827. PMID 18509334.
- ^ Hattori, M .; и другие. (2009). «Диагностика синдрома Рассела-Сильвера с помощью комбинированного анализа бисульфитной рестрикции-денатурирующей высокоэффективной жидкостной хроматографии». Генетическое тестирование и молекулярные биомаркеры. 13 (5): 623–630. Дои:10.1089 / gtmb.2009.0018. PMID 19814617.
- ^ Фрага, Марио Ф .; Эстеллер, Манель (2002). «Метилирование ДНК: профиль методов и приложений». Биотехнологии. 33 (3): 633–649. Дои:10.2144 / 02333rv01. PMID 12238773.

