ЧИА-ПЭТ - ChIA-PET
Анализ взаимодействия хроматина с помощью секвенирования парных концевых меток (ЧИА-ПЭТ или же ЧИА-ПЕТС) - это метод, включающий иммунопреципитация хроматина (ChIP) -обогащение, лигирование близости хроматина, Парные теги, и Секвенирование с высокой пропускной способностью определить de novo дальний хроматин взаимодействия в масштабе всего генома.[1]
Гены возможно регулируемый участками, удаленными от промотора, такими как регуляторные элементы, инсуляторы и граничные элементы, и сайты связывания факторов транскрипции (TFBS). Раскрытие взаимодействия между регулирующий регионы и кодирующие области генов необходим для понимания механизмов, управляющих генная регуляция в здоровье и болезнь (Мастон и др., 2006). ChIA-PET можно использовать для идентификации уникальных функциональных хроматин взаимодействие между дистальным и проксимальным регулирующий сайты связывания факторов транскрипции и промоутеры генов, с которыми они взаимодействуют.
ChIA-PET также может быть использован для раскрытия механизмов геном контроль во время таких процессов, как дифференциация клеток, распространение, и разработка. Создав ЧИА-ПЭТ интерактом карты для ДНК-связывающих регуляторных белков и промоутер регионах, мы можем лучше определить уникальные цели для терапевтического вмешательства (Fullwood & Yijun, 2009).
Методология
Метод ChIA-PET объединяет методы на основе ChIP (Kuo & Allis, 1999) и Захват конформации хромосомы (3C), чтобы расширить возможности обоих подходов. ChIP-Sequencing (ChIP-Seq) - популярный метод, используемый для идентификации TFBS, тогда как 3C используется для идентификации дальнодействующих взаимодействий хроматина (Dekker et al., 2002). Тем не менее, оба имеют ограничения при использовании независимо друг от друга для выявления дальнодействующих взаимодействий de novo в масштабе всего генома. Хотя ChIP-Seq обычно используется для полногеномной идентификации TFBS (Barski et al., 2007; Wei et al., 2006), он предоставляет только линейную информацию о сайтах связывания белков вдоль хромосом (но не о взаимодействиях между ними), и может страдать от высокого геномного фонового шума (ложные срабатывания).
Хотя 3C способен анализировать дальнодействующие взаимодействия хроматина, он не может использоваться в масштабах всего генома и, как ChIP-Seq, также страдает от высоких уровней фонового шума. Поскольку шум увеличивается в зависимости от расстояния между взаимодействующими регионами (макс. 100kb), требуются трудоемкие и утомительные средства контроля для точной характеристики взаимодействий хроматина (Dekker et al., 2006).
Метод ChIA-PET успешно решает проблемы неспецифического взаимодействия шума, обнаруженного в ChIP-Seq, путем обработки фрагментов ChIP ультразвуком, чтобы отделить случайные присоединения от определенных комплексов взаимодействия. Следующий шаг, который называется обогащением, снижает сложность анализа в масштабе всего генома и добавляет специфичность взаимодействиям хроматина, связанным с заранее определенными TF (факторами транскрипции). Способность подходов 3C определять дальнодействующие взаимодействия основана на теория близости перевязки. Что касается интерлигирования ДНК, фрагменты, которые связаны общими белковыми комплексами, имеют большие кинетические преимущества в условиях разбавления, чем фрагменты, свободно диффундирующие в растворе или закрепленные в различных комплексах. ChIA-PET использует преимущества этой концепции путем включения линкерных последовательностей на свободные концы фрагментов ДНК, связанных с белковыми комплексами. Для создания связности фрагментов, связанных регуляторными комплексами, линкерные последовательности лигируют во время лигирования ядерной близости. Таким образом, продукты лигирования с линкерным соединением могут быть проанализированы с помощью секвенирования ПЭТ со сверхвысокой пропускной способностью и сопоставлены с эталонный геном. Поскольку ChIA-PET не зависит от специфических сайтов для обнаружения, как 3C и 4C, он позволяет беспристрастное, общегеномное обнаружение взаимодействий хроматина de-novo (Fullwood et al., 2009).
Рабочий процесс
Часть рабочего процесса влажной лаборатории:



- Формальдегид используется для сшивания комплексов ДНК-белок. Обработка ультразвуком используется для разрушения хроматина, а также для уменьшения неспецифических взаимодействий.
- Выбранное специфическое антитело используется для обогащения фрагментов хроматина, связанных с представляющим интерес белком. Материал ChIP, связанный с антителом, используется для создания ChIA-PET.
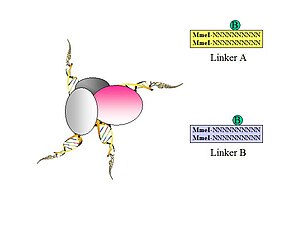
- Фигура 1. Биотинилированные олигонуклеотидные полулинкеры, содержащие фланкирующие сайты MmeI, используются для соединения близких лигированных фрагментов ДНК. Разработаны два разных линкера (A и B) со специфическими нуклеотидными штрих-кодами (CG или AT) для каждой из двух линкерных последовательностей (это позволит идентифицировать химерный продукт лигирования, как показано на рисунке 5.).
- Рисунок 2. Линкеры лигированы к привязанным фрагментам ДНК.
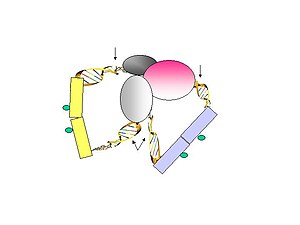
- Фигура 3. Фрагменты линкера лигируют на гранулы ChIP в разбавленных условиях. Очищенная ДНК затем переваривается MmeI, который разрезает на расстоянии от своего сайта узнавания, чтобы высвободить структуру метка-линкер-метка.
- Рисунок 4. Биотинилированные ПЭТ затем иммобилизуют на магнитных шариках, конъюгированных со стрептавидином.
- Фигура 5. Последовательности ПЭТ с композицией линкерного штрих-кода AA (CG / CG) и BB (AT / AT) считаются возможными продуктами внутрикомплексного лигирования, в то время как последовательности ПЭТ с композицией линкера AB (CG / AT) считаются возможными продуктами лигирования. происходит из химерных продуктов лигирования между фрагментами ДНК, связанными в различные комплексы хроматина.
Лабораторная часть рабочего процесса:
Извлечение ПЭТ, картирование и статистический анализ
Теги ПЭТ извлекаются и сопоставляются с эталонным геномом человека. in silico.
Идентификация пиков, обогащенных ChIP (сайтов связывания)
Самолигирующийся ПЭТ используется для идентификации сайтов, обогащенных ChIP, поскольку они обеспечивают наиболее надежное отображение (20 + 20 бит / с) на эталонный геном.
Алгоритм поиска пиков обогащения ChIP
Вызванный пик считается сайтом связывания, если имеется несколько перекрывающихся самолигируемых ПЭТ. Уровень ложного обнаружения (FDR) определяется с помощью статистического моделирования для оценки случайного фона перекрытий виртуальной ДНК, полученных из ПЭТ, и оцененного фонового шума.
Фильтрация повторяющейся ДНК (влияет на неспецифическое связывание)
Спутниковые области и сайты связывания, присутствующие в регионах с серьезными структурными вариациями, удаляются.
Подсчет обогащения ChIP
Количество ПЭТ с самолигированием и интерлигированием (в пределах окна + 250 п.н.) сообщается на каждом сайте. Общее количество самолигированных и интерлигированных ПЭТ в определенном месте называется подсчетом обогащения ChIP.
Рисунок 6. Классификация ПЭТ: уникально выровненные последовательности ПЭТ можно классифицировать по тому, происходят ли они из одного фрагмента ДНК или двух фрагментов ДНК.

- Самолигирование ПЭТ
Если два тега ПЭТ картированы на одной и той же хромосоме с геномным диапазоном в диапазоне фрагментов ДНК ChIP (менее 3 КБ), с ожидаемой ориентацией самолигирования и на одной и той же цепи, они считаются производными от самолигирование одного фрагмента ДНК ChIP и рассматривается как самолигирование ПЭТ.
- ПЭТ с интерлигированием
Если ПЭТ не соответствует этим критериям, то ПЭТ, скорее всего, является результатом лигирования двух фрагментов ДНК и называется ПЭТ между лигированием. Два тега ПЭТ с интерлигированием не имеют фиксированной ориентации тегов, могут не находиться на одних и тех же цепях, могут иметь любой геномный диапазон и могут не отображаться на одной и той же хромосоме.
- Внутрихромосомные интерлигированные ПЭТ
Если два тега межлигированного ПЭТ картированы в одной и той же хромосоме, но с размахом> 3 Кбайт в любой ориентации, то эти ПЭТ называются внутрихромосомными интерлигативными ПЭТ.
- Межхромосомные интерлигированные ПЭТ
ПЭТ, которые сопоставлены с разными хромосомами, называются ПЭТ с межхромосомным интерлигированием.
Рисунок 7. Предлагаемый механизм, показывающий, как дистальные регуляторные элементы могут инициировать дальнодействующие взаимодействия хроматина с участием промоторных областей генов-мишеней.

Взаимодействия образуют петлевые структуры ДНК с множеством TFBS в центре закрепления. Маленькие петли могут упаковывать гены рядом с центром закрепления в плотный субкомпартмент, что может увеличивать локальную концентрацию регуляторных белков для усиленной активации транскрипции. Этот механизм может также повышать эффективность транскрипции, позволяя РНК pol II циклировать плотные кольцевые генные матрицы. Большие петли взаимодействия с большей вероятностью будут связывать вместе удаленные гены на любом конце петли, расположенные рядом с якорными сайтами для скоординированной регуляции, или могут разделять гены в длинные петли, чтобы предотвратить их активацию. По материалам Fullwood et al. (2009).
Сильные и слабые стороны
Преимущества метода ChIA-PET
- ChIA-PET потенциально может быть беспристрастным, полногеномным и новым подходом для анализа взаимодействия хроматина на больших расстояниях (Fullwood & Yijun, 2009).
- Эксперимент ChIA-PET может предоставить два глобальных набора данных: сайты связывания белкового фактора (самолигирующиеся PET); и Взаимодействия между сайтами связывания (интерлигированные ПЭТ).
- ChIA-PET включает ChIP, чтобы упростить общегеномный анализ и добавить специфичность взаимодействиям хроматина, связанным с конкретными интересующими факторами.
- ChIA-PET совместим с подходами к секвенированию следующего поколения на основе тегов, такими как пиросеквенирование Roche 454, Illumina GA, ABI SOLiD и Helicos.
- ChIA-PET применим ко многим различным белковым факторам, участвующим в регуляции транскрипции или структурной конформации хроматина.
- Анализ ChIA-PET может быть применен к взаимодействиям хроматина, вовлеченным в конкретный ядерный процесс. Используя общие TF, такие как РНК-полимераза II, можно идентифицировать все взаимодействия хроматина, участвующие в регуляции транскрипции. Более того, использование белковых факторов, участвующих в репликации ДНК или структуре хроматина, позволит идентифицировать все взаимодействия, обусловленные репликацией ДНК и структурной модификацией хроматина (Fullwood et al., 2009).
Недостатки
- Хорошо известно, что цис- и транс-регуляторные комплексы содержат уникальные комбинации белков, основанные на условиях, специфичных для клеток и тканей (Dekker et al., 2006). Хотя идентификация одиночного функционального TFBS является значительным достижением, использование ChIA-PET для идентификации отдельных белков в комплексе потребует работы наугад и нескольких экспериментов для идентификации каждого взаимодействующего белка. Это будет дорогостоящим и трудоемким мероприятием.
- ХИА-ПЭТ ограничен качеством, чистотой и специфичностью используемых антител (Fullwood et al., 2009).
- ChIA-PET зависит от идентификации последовательностей, которые могут быть сопоставлены с эталонной последовательностью (ссылка).
- ChIA-PET требует использования компьютерных алгоритмов пикового вызова для организации и сопоставления считываний ПЭТ с эталонным геномом. Из-за различий между программными платформами результаты могут отличаться в зависимости от того, какая программа используется.
- Хотя повторяющиеся участки ДНК могут быть связаны с регуляцией генов (Polak & Domany, 2006), они должны быть удалены, поскольку они могут влиять на данные (Fullwood et al., 2009).
История
Fullwood et al. (2009) использовали ChIA-PET для обнаружения и картирования сети взаимодействия хроматина, опосредованной рецептор эстрогена альфа (ER-альфа) в раковых клетках человека. Полученная в результате глобальная карта взаимодействия хроматина показала, что удаленные сайты связывания ER-альфа также были прикреплены к промоторам генов посредством дальнодействующих взаимодействий хроматина, предполагая, что ER-альфа функционирует посредством обширного образования петель хроматина, чтобы объединить гены для координированной регуляции транскрипции.
Анализ и программное обеспечение
Программное обеспечение, обычно используемое в эксперименте ChIA-PET
ELAND
Сопоставляет фрагменты ДНК, обогащенные ChIP, с эталонным геномом человека.[1]
RepeatMasker
In-silico маскирование повторяющихся элементов.[2]
Моделирование Монте-Карло
Используется для оценки вероятности ложного обнаружения.[2]
ПЭТ-инструмент
Программный пакет для обработки и управления данными последовательностей парных ди-тегов.[3]
Инструмент ChIA-PET
Программный комплекс для обработки данных ChIA-PET.Веб-сайт ChIA-PET Tool ChIA-PET Инструментальная бумага
Альтернативы
Иммунопреципитация хроматина:
ChIP-Seq, ЧИП-ПЭТ, ЧИП-ШАЛФЕЙ, ЧИП-ЧИП.
2-С, 3-С, 4-С, 5-С.
Парные конечные теги:
Рекомендации
- ^ Фуллвуд, Мелисса Дж .; Жуань Ицзюнь (2009-05-01). «Методы на основе ChIP для идентификации дальнодействующих взаимодействий хроматина». Журнал клеточной биохимии. 107 (1): 30–39. Дои:10.1002 / jcb.22116. ISSN 1097-4644. ЧВК 2748757. PMID 19247990.
- ^ Метод Монте-Карло
- Барски и др., (2007). Профилирование метилирования гистонов в геноме человека с высоким разрешением. Клетка. (129); 823–37.
- Деккер, (2002). Захват конформации хромосомы. Наука. (295); 1306–1311.
- Деккер, (2006). Три «C» конформации хромосомы: контроль, контроль, контроль. Nat. Методы. (3); 17–21.
- Fullwood et al., (2009). Связанный с эстроген-рецептором-α хроматин человека взаимодействует. Природа. (462); 58–64.
- Фулвуд и Иджун, (2009). Методы на основе ChIP для идентификации дальнодействующих взаимодействий хроматина. J Cell Biochem. 107 (1); 30–39.
- Джонсон и др., (2007). Полногеномное картирование взаимодействий белок-ДНК in vivo. Наука. (316); 1497–502.
- Куо и Аллис, (1999). Сшивание in vivo и иммунопреципитация для изучения динамических ассоциаций белок: ДНК в среде хроматина. Методы. (19); 425–33.
- Ли, Дж., Фулвуд, MJ, Сюй, Х., Мулавади, Ф.Х., Велков, С., Вега, В., Арияратне, П.Н., Мохамед, YB, Оои, HS, Теннакун, К., Вэй, CL, Руан , Ю. и Сун, В.К. Инструмент ChIA-PET для всестороннего анализа взаимодействия хроматина с секвенированием парных концевых меток. Геном Биол, 11 (2). R22.
- Мастон и др., (2006). Элементы регуляции транскрипции в геноме человека. Анну. Преподобный: Геномика. Hum Genet. (7); 29–59.
- Полак и Доманы, (2006). Элементы Alu содержат множество сайтов связывания для факторов транскрипции и могут играть роль в регуляции процессов развития. BMC Genomics. (7); 133.
- Wei et al., (2006). Глобальная карта сайтов связывания фактора транскрипции p53 в геноме человека. Клетка. (124); 207–19.
внешняя ссылка
- Браузер генома ChIA-PET - Этот браузер предназначен для просмотра данных Fullwood et al. (2009), и включает в себя настраиваемую программу просмотра взаимодействия всего генома, которая обеспечивает макроскопическую картину участков связывания и взаимодействий вместе с ландшафтом всего генома.
